NanoString nCounter Nmax
En nuestra Plataforma de Detección Molecular Directa tenemos novedades que contaros:
Nuestro recién adquirido equipamiento nCounter MAX de Nanostring ofrece una serie de ventajas sobre la versión anterior (SPRINT Profiler), y nos permite mejorar tiempos de análisis eliminando los principales cuellos de botella en el procesado de muestras y obtención de datos y resultados para tú proyecto o estudio. Además, permite detectar pequeños cambios estructurales en el plegamiento. Elimina la síntesis, amplificación y preparación de bibliotecas de cDNA para reducir variaciones técnicas en el ensayo y reduce la necesidad de réplicas experimentales.
El sistema de análisis nCounter® permite analizar cientos de mRNAs, miRNAs, SNV o CNV directamente mediante detección molecular digital directa, en una sola reacción en ausencia de enzimas (sin retrotranscripción ni amplificación). Es un sistema de alta sensibilidad y reproducibilidad con gran capacidad de multiplexado (hasta 800 genes en la misma reacción), lo cual no solo disminuye el número de reacciones necesarias, sino que también supone un ahorro en la cantidad de RNA/DNA que se requiere para el ensayo.
Tecnología
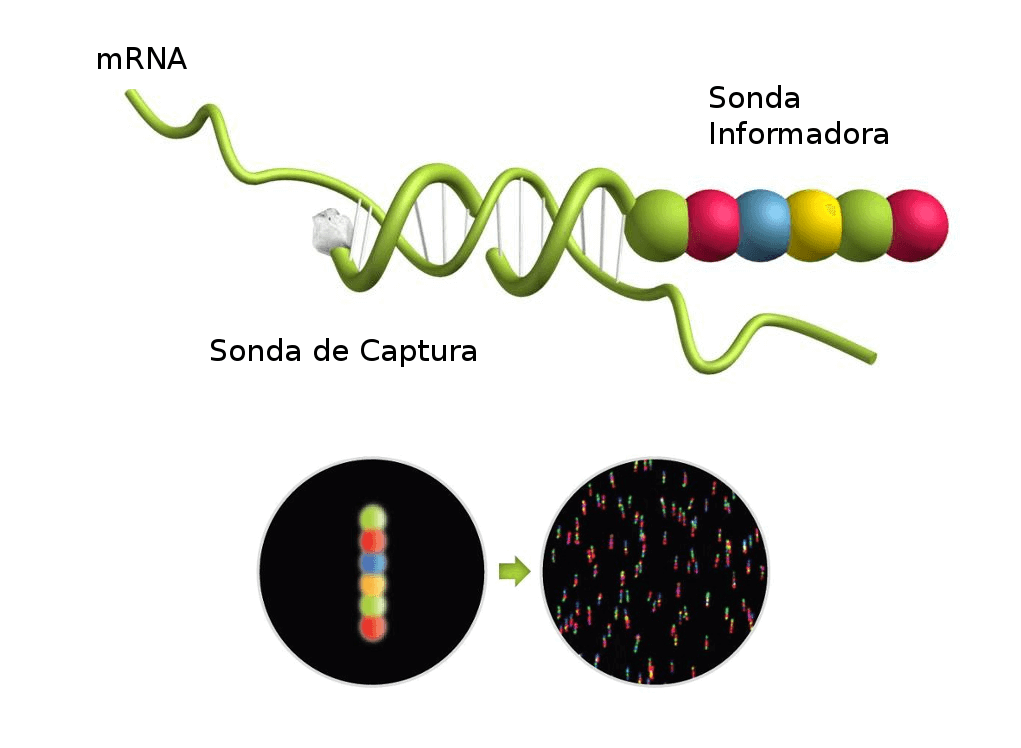
En esta tecnología, cada molécula diana viene identificada por un “código de barras” molecular formado por la combinación secuencial de seis unidades de fluorocromos de cuatro colores distintos.
Estos códigos de barras hibridan directamente con las moléculas de interés, y pueden ser cuantificadas individualmente sin ser necesaria la amplificación de las muestras, proporcionando así datos digitales con una gran sensibilidad.
Cada “Código de Barras” se une a una molécula diana individual y específica, de manera que logramos medir la cantidad real de moléculas diana existentes en una muestra.


Ventajas de la Tecnología
- Rapidez: No requiere de ningún tipo de reacción enzimática (ni amplificación, obtención de cDNA ni preparación de librerías). Por lo tanto, evita la variación de tipo técnico y, por consiguiente, un posible sesgo en los resultados.
- Requiere poca cantidad de RNA: por lo que el ahorro de muestra es considerable.
- Alta precisión, sensibilidad y reproducibilidad: precisión equiparable a la de una qPCR y sin necesidad de hacer réplicas. La correlación entre réplicas técnicas excede 0.99. La precisión y sensibilidad se mantiene incluso con niveles bajos de expresión.
- Versatilidad: la química es altamente tolerante a tipos de muestra difíciles. Al no requerir enzimas, el sistema puede trabajar con una gran variedad de tipos y calidades de muestras, siendo posible incluso obtener resultados óptimos a partir de muestras parafinadas (FFPE) donde el material de partida está muy degradado, o incluso puede trabajar con lisados celulares sin necesidad de llevar a cabo una extracción de ARN.
- Capacidad de multiplexado: analiza cientos de targets en un solo tubo (hasta 800 genes). Posibilidad de usar paneles pre-diseñados o construir paneles custom a medida con las dianas de interés
- Tratamiento de datos sencillo: salida de datos en formato csv. Los datos pueden ser exportados y explotados con softwares de uso habitual.
Aplicaciones
Expresión Génica
La tecnología tiene un gran número de aplicaciones potenciales en diferentes ámbitos de la investigación clínica a través de los numerosos paneles pre-diseñados (12 muestras) que están disponibles para la plataforma, así como a través de la posibilidad de diseñar paneles personalizados a medida del usuario con los targets de interés (hasta 800 dianas).
-
Paneles pre-diseñados relacionados con el estudio del cáncer:
- Paneles de expresión que contienen aquellos genes implicados en la formación de procesos oncogénicos.
- Paneles de expresión que contienen aquellos genes relacionados con la respuesta inmune en diferentes tipos de cáncer.
- Paneles de expresión que contienen genes implicados en la progresión de un tumor.
- Paneles para el estudio de genes de fusión enfocado principalmente al diagnóstico de ciertos tumores.
- Paneles pre-diseñados relacionados con la expresión de genes implicados en rutas inmunológicas, inflamatorias y neurodegenerativas tanto en humanos como en ratón y en rata
- Tecnología PlexSet: Multiplexado de muestras. Panel a medida de hasta 96 genes y 96 muestras por cartucho (configuraciones de 12, 24, 48, 72 y 96 genes). Solución coste efectiva y perfecta para validación de firmas.
-
miRNAs:
- Panel de miRNAs con 800 miRNAs más relevantes de funcionalidad estrictamente biológica. Descubrimiento y validación de miRNAs en un solo panel.
- Panel miRGE personalizado capaces de evaluar simultáneamente mRNAs y miRNAs en la misma muestra.
- lncRNA: Panel a medida de hasta 800 lncRNA. Pueden combinarse con mRNA para análisis simultáneo o para estudiar interacciones lncRNA-proteínas (Inmunoprecipitación).
- Paneles para estudios de expresión de célula única con una gran variedad de tipos de muestra de partida: sorting, microdisección o micromanipulación.
- Nuevos paneles pre-diseñados para enfermedades cardiovasculares, terapia celular y génica y células madre.
DNA
- Análisis de Variación de Número de Copias (CNV). Detección y análisis de hasta 800 regiones diana en una sola reacción. Elevada precisión en CNVs multialélicos. Posibilidad de trabajar con muestras parafinadas. Reproducibilidad mayor que las plataformas de microarrays (R2 = 0.99 en el caso de NanoString frente a un R2 =0.91 en microarrays).
- ChIP-String: Estudio de como el DNA interacciona con las proteínas para regular la expresión génica. Excelente opción tanto para validar los resultados obtenidos por ChIP-Seq como para estudiar loci implicados en la remodelación de la cromatina y la expresión génica en un número grande de muestras.
Proteómica
El análisis de proteínas NanoString aprovecha anticuerpos específicos marcados con códigos de barras de colores a través de oligonucleótidos únicos de ADN sintético para la detección de proteínas de interés. La detección de proteínas con esta metodología correlaciona fuertemente con ensayos de citometría de flujo (R20>.75).
Nanostring permite la cuantificación simultánea y en la misma muestra de proteínas y genes de expresión haciendo uso de la misma tecnología con los nCounter® Vantage 3D™ Immune Assays. Estos ensayos permiten analizar el mRNA y las proteínas de superficie celular como receptores, marcadores de linaje celular, moléculas reguladoras para monitorizar cambios en la abundancia de un determinado tipo celular y su status de activación. También permite estudiar el mRNA y los niveles de determinadas proteínas intracelulares como las citoquinas, quimoquinas y factores de transcripción implicados en estadios iniciales de comunicación celular y diferenciación inmune.
Servicio Nanostring de GENvip
El servicio ofrecido por GENvip incluye
Soporte
Soporte y asesoramiento con el diseño del experimento y desarrollo del protocolo.
Material
Compra de reactivos, CodeSets y cartuchos necesarios para llevar a cabo el experimento.
Análisis
Preparación y procesado de 12 muestras por cartucho, hibridación y análisis en el equipo nCounter MAX.
Resultados
Archivo CSV con los datos crudos exportados del equipo y datos normalizados. Asesoramiento con el análisis de datos
Muestras
Las muestras pueden ser extraídas de tejidos frescos, líneas celulares o parafinas. Se pueden emplear RNA total, miRNA, DNA o lisados crudos dependiendo del tipo de proyecto. Número mínimo de muestras son 12.
- Calidad de la muestra: el material genético debe estar libre de contaminantes (Pureza: A260/A280 ratio de 1.7–2.3 y A260/A230 ratio de 1.8–2.3) y el 50% de los fragmentos debería estar por encima de 300pb.
- Cantidad de muestra:
- mRNA: Mínimo de 100ng de mRNA total dependiendo del panel (concentración mínima 20ng/µl)
- miRNA: 100-300ng de mRNA total (concentración mínima de 33ng/µl)
- CNV y ChIP-String: 200-600ng de DNA (concentración mínima 29 ng/µl)
El grupo GENVIP es un equipo multidisciplinar de naturaleza transversal que engloba diferentes disciplinas en el ámbito de la investigación clínica y básica. GENVIP emplea las últimas tecnologías y métodos analíticos de las diferentes disciplinas para optimizar el tratamiento de los pacientes pediátricos.
El objetivo principal del grupo de investigación GENVIP es profundizar en el conocimiento de la patogénesis de las enfermedades infecciosas que afectan a niños desde diferentes perspectivas, a través de una aproximación “ómica”. Esta estrategia nos llevará a identificar biomarcadores genéticos y transcritptómicos que nos permitirán definir firmas asociadas al diagnóstico o pronóstico de las diferentes patologías infecciosas pediátricas basándonos en su fenotipo molecular. Esto proporcionará información que podrá ser empleada de forma traslacional en la práctica clínica, contribuyendo al diagnóstico temprano, prevención selectiva y tratamiento de los pacientes pediátricos. Este conocimiento también abrirá nuevas líneas para el estudio de tratamientos personalizados de los pacientes, contribuyendo al entendimiento y la predicción del impacto de los productos terapéuticos y las vacunas en el sistema inmune del niño.





















